Es ist ja eigentlich ganz einfach:
Zählt man die Infektionen und Toten ab dem ersten weltweiten Ausbruch der Pandemie, dann müssen alle Zahlen, auch die aus China, Italien, USA, Indien usw. berücksichtigt werden.
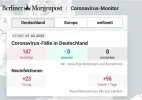
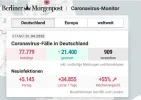
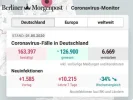
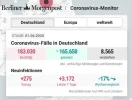
Will man sich nur mit den Zahlen in Deutschland befassen, so schaut man, ab wann sich der Erreger in D. so richtig ausgebreitet hat.
Den ersten Corona-Toten in D. gab es jedenfalls erst Anfang März 2020.
ok, dann müsste man den unterschied ja schon zwischen den monaten januar, februar und märz, april, mai usw schon im jahre 2020 erkennen.
hier mal eine übersicht der monate von 2016 bis 2020. an welchen zahlen ekennt man genau diese coronabedingte übersterblichkeit?





dass die koch'schen postulate nicht mehr gelten stimmt übrigens nicht. sie wurden nur in einem der punkte abgewandelt,
weil es manche viren geben soll die sich nicht isolieren lassen.
dafür wird dann die jedem sofort leicht verständliche

Metatranskriptomanalyse angewendet:
Werkzeuge und Techniken Obwohl Microarrays zur Bestimmung der Genexpressionsprofile einiger Modellorganismen genutzt werden können, sind Sequenzierung der nächsten Generation und Sequenzierung der dritten Generation die bevorzugten Techniken in der Metatranskriptomik. Das Protokoll, das zur Durchführung einer Metatranskriptomanalyse verwendet wird, kann je nach Art der zu analysierenden Probe variieren. In der Tat wurden viele verschiedene Protokolle entwickelt, um das Metatranskriptom von mikrobiellen Proben zu untersuchen. Im Allgemeinen umfassen die Schritte Probenentnahme, RNA-Extraktion (verschiedene Extraktionsmethoden für verschiedene Arten von Proben wurden in der Literatur beschrieben), mRNA-Anreicherung, cDNA-Synthese und Herstellung von metatranskriptomischen Bibliotheken, Sequenzierung sowie Datenverarbeitung und -analyse. Die mRNA-Anreicherung ist einer der schwierigsten Teile. Es wurden verschiedene Strategien vorgeschlagen: Entfernen von rRNA durch Einfangen von ribosomaler RNA unter Verwendung einer 5-3-Exonuklease zum Abbau verarbeiteter RNAs (meist rRNA und tRNA ) Zugabe von Poly (A) zu mRNAs unter Verwendung einer PolyA-Polymerase (in E. coli ) Verwendung von Antikörpern zum Einfangen von mRNAs, die an bestimmte Proteine binden Die letzten beiden Strategien werden nicht empfohlen, da berichtet wurde, dass sie stark voreingenommen sind. Metatranskriptomik -
https://de.qaz.wiki/wiki/Metatranscriptomics
Computeranalyse Eine typische Pipeline zur Metatranskriptomanalyse: Karten liest zu einem Referenzgenom oder führt die De-novo-Assemblierung der Lesevorgänge zu Transkript-Contigs und Supercontigs durch Die erste Strategie ordnet Lesevorgänge Referenzgenomen in Datenbanken zu, um Informationen zu sammeln, die nützlich sind, um die relative Expression der einzelnen Gene abzuleiten. Metatranskriptomische Lesevorgänge werden mithilfe von Ausrichtungswerkzeugen wie Bowtie2 , BWA und BLAST auf Datenbanken abgebildet . Anschließend werden die Ergebnisse mit Ressourcen wie GO , KEGG , COG und Swiss-Prot kommentiert . Die endgültige Analyse der Ergebnisse erfolgt je nach Ziel der Studie. Eine der neuesten Metatranskriptomik-Techniken ist die stabile Isotopensuche (SIP), mit der spezifische zielgerichtete Transkriptome aerober Mikroben in Seesedimenten gewonnen werden. Die Einschränkung dieser Strategie besteht darin, dass sie sich auf die Informationen von Referenzgenomen in Datenbanken stützt. Die zweite Strategie ermittelt die Häufigkeit der Expression der verschiedenen Gene, indem metatranskriptomische Lesevorgänge unter Verwendung verschiedener Software zu längeren Fragmenten, sogenannten Contigs, zusammengesetzt werden. Die Grenzen hängen also von der Software ab, die für die Baugruppe verwendet wird. Es wurde berichtet, dass die Trinity-Software für RNA-seq im Vergleich zu anderen De-novo-Transkriptom-Assemblern über einen breiten Bereich von Expressionsniveaus mehr Transkripte voller Länge mit einer Empfindlichkeit gewinnt, die Methoden ähnelt, die auf Genom-Alignments beruhen. Dies ist besonders wichtig, wenn kein Referenzgenom vorhanden ist. Eine quantitative Pipeline für die Transkriptomanalyse wurde von Li und Dewey entwickelt und als RSEM (RNA-Seq by Expectation Maximization) bezeichnet. Es kann als eigenständige Software oder als Plug-In für Trinity verwendet werden. RSEM beginnt mit einem Referenztranskriptom oder einer Referenzassemblierung zusammen mit aus der Probe generierten RNA-Seq-Lesevorgängen und berechnet die normalisierte Transkripthäufigkeit (dh die Anzahl der RNA-Seq-Lesevorgänge, die jedem Referenztranskriptom oder jeder Referenzassemblierung entsprechen). Obwohl sowohl Trinity als auch RSEM für transkriptomische Datensätze entwickelt wurden (dh von einem einzelnen Organismus erhalten wurden), können sie möglicherweise auf metatranskriptomische Daten angewendet werden (dh von einer ganzen mikrobiellen Gemeinschaft erhalten). Metatranskriptomik -
https://de.qaz.wiki/wiki/Metatranscriptomics
https://de.qaz.wiki/wiki/Metatranscriptomics
mit dieser technik haben chinesen im januar 2020 die erste sequenzierung bekannt gegeben, auf die sich drosten
für seinen test stütze:
"Man habe, so Drosten, eine Reihe von Tests auf Basis des ersten SARS-Virus gemacht, die eine „riesengroße Diversität von Fledermaus-Coronaviren, also [den] nächsten Verwandten“ beinhalteten. Als in China eine
Sequenzinformation des neuen Coronavirus veröffentlicht wurde, habe man die Daten mit den Tests abgeglichen. Es seien zwei Tests ausgewählt worden, die am besten zu dem neuen Virus passten. "
https://correctiv.org/faktencheck/2...-wurde-isoliert-und-pcr-tests-sind-validiert/
und hier zu der sequenzinformation aus china:
https://archive.is/pSYah